


 В биомедицинских исследованиях алгоритмы машинного обучения часто используются для анализа данных, например для предсказания рецидива рака. Однако не всегда ясно, находят ли эти алгоритмы значимые закономерности или подстраиваются под случайные шумы в данных. Ученые из НИУ ВШЭ, ИБХ РАН и МГУ разработали тест, который позволяет определить эту разницу. Он может стать важным инструментом для проверки надежности алгоритмов в медицине и биологии. Исследование опубликовано в цифровом архиве arXiv.
В биомедицинских исследованиях алгоритмы машинного обучения часто используются для анализа данных, например для предсказания рецидива рака. Однако не всегда ясно, находят ли эти алгоритмы значимые закономерности или подстраиваются под случайные шумы в данных. Ученые из НИУ ВШЭ, ИБХ РАН и МГУ разработали тест, который позволяет определить эту разницу. Он может стать важным инструментом для проверки надежности алгоритмов в медицине и биологии. Исследование опубликовано в цифровом архиве arXiv.
Методы машинного обучения помогают анализировать сложные биологические данные, например предсказывать вероятность рецидива рака по экспрессии генов — уровню активности участков ДНК в клетках. Однако не всегда ясно, находят ли эти алгоритмы значимые закономерности или подстраиваются под случайные шумы в данных.
Команда ученых из НИУ ВШЭ, ИБХ РАН и МГУ разработала тест, который позволяет проверить, насколько надежно классификатор различает группы пациентов. В данном случае речь идет о двух группах: те, у кого рецидив произошел, и те, у кого его не было. Если модель действительно выявляет биологически значимые различия, значит, она работает корректно. Если же алгоритм просто случайно делит данные, его точность может быть обманчиво высокой. Ученые сосредоточились на линейных классификаторах — одном из самых частых инструментов машинного обучения, применяемых в биомедицине.
« Мы хотели проверить, насколько вероятно, что даже случайно сгенерированные (синтетические) данные можно разделить линейным классификатором не хуже, чем реальные биологические образцы. Для этого мы рассчитали верхнюю границу p-значения — число, которое показывает вероятность того, что модель "угадывает". Чем ниже это значение, тем надежнее классификатор. »
Антон Жиянов
научный сотрудник Лаборатории молекулярной физиологии НИУ ВШЭ
Исследователи провели серию экспериментов на синтетических данных, в ходе которых могли точно контролировать степень различий между классами. Затем они применили новый тест к реальным медицинским моделям, предсказывающим риск рецидива рака молочной железы.
Оказалось, что большинство классификаторов не выявляли реальных различий между пациентами с рецидивом и без него. При дополнительной проверке 559 из 570 моделей показали случайные результаты. Это значит, что многие алгоритмы могут казаться точными, хотя на самом деле их предсказания основаны на совпадениях, а не на реальных закономерностях.
Однако исследователи нашли и надежные модели, которые выявляют биологически значимые закономерности. Одной из них оказался классификатор, который ориентировался на уровень активности генов ELOVL5 и IGFBP6. Этот алгоритм прошел дополнительную проверку на независимой выборке данных и показал, что различия в экспрессии этих генов действительно связаны с риском рецидива рака.
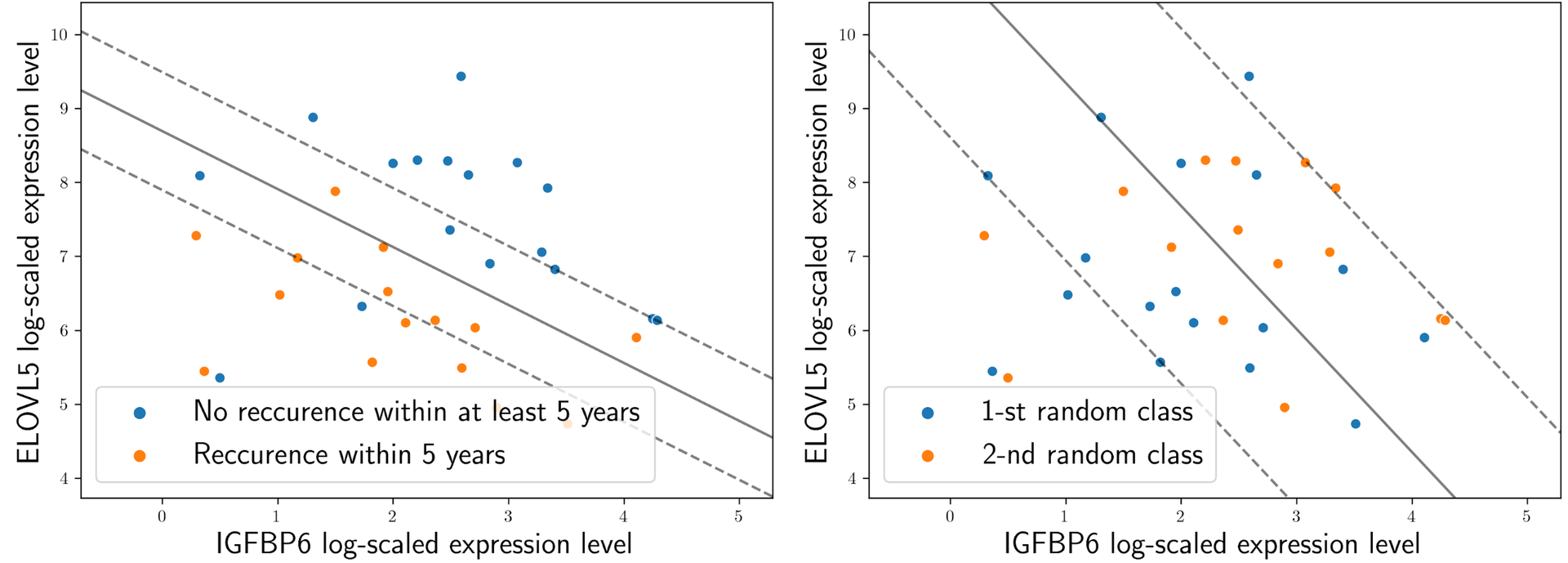
Рис. 1. Первый график — «неслучайный классификатор», основанный на экспрессии IGFBP6 и ELOVL5; второй — случайный классификатор. ©Zhiyanov A. et al. Statistical Verification of Linear Classifiers //arXiv preprint arXiv:2501.14430. – 2025.
Каждая точка на графике — человек, у которого измерили экспрессию двух генов — IGFBP6 (по оси X) и ELOVL5 (по оси Y). Оранжевые точки — люди с рецидивом, а синие — без. На первом графике эти точки (люди) четко разделены прямой (линейным классификатором). На втором графике точки расположены хаотично, классификатор не идентифицирует закономерности между экспрессией и реальным рецидивом.
« Наш тест может стать важным инструментом для проверки надежности алгоритмов в биологии и медицине. Он помогает избежать ложных выводов и сосредоточиться на моделях, которые действительно находят важные закономерности, что критично для принятия решений о лечении пациентов. »
Александр Тоневицкий
профессор факультета биологии и биотехнологии НИУ ВШЭ
Работа выполнена при поддержке Программы фундаментальных исследований НИУ ВШЭ в рамках проекта «Центры превосходства».
Все выпуски журнала «ЭкоГрад» в электронной версии читайте на pressa.ru,
Бумажные экземпляры спецвыпусков и книги В. Климова можно приобрести на OZON